Un exbecario de investigación postdoctoral de la Universidad de la Columbia Británica (UBC) dirigió un equipo de investigación internacional para volver a analizar todos los datos públicos de secuenciación de ARN, con el fin de descubrir casi diez veces más virus de ARN de los que se conocían anteriormente, incluidas varias especies nuevas de coronavirus en algunos lugares inesperados.
Esta base de datos a escala planetaria de virus de ARN puede ayudar a allanar el camino para identificar rápidamente la propagación del virus a los humanos, así como los que afectan al ganado, los cultivos y las especies en peligro de extinción.
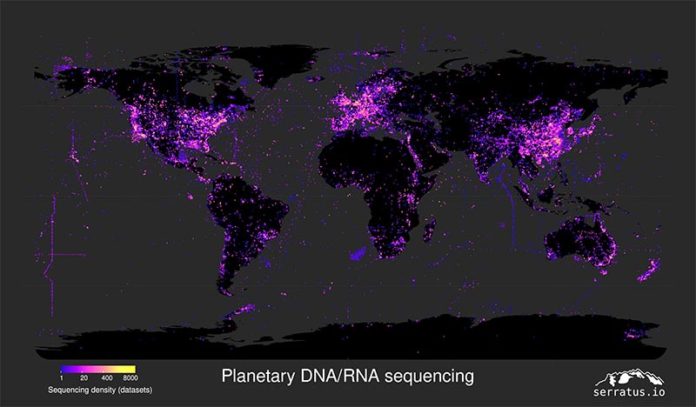
20 millones de gigabytes de 5,7 millones de muestras biológicas en busca de virus de ARN
El Dr. Artem Babaian está detrás de la colaboración del Proyecto Serratus, y publicó los sorprendentes resultados de la investigación en la revista Nature esta semana.
Trabajando con el Centro de Innovación en la Nube, una colaboración pública/privada entre la UBC y Amazon Web Services, el Proyecto Serratus pudo construir un superordenador «ridículamente potente» en AWS con una potencia equivalente a 22.500 CPU, según Babaian.
El superordenador leyó 20 millones de gigabytes de datos de secuencias de genes disponibles públicamente de 5,7 millones de muestras biológicas en todo el mundo, en busca de un gen específico que indicara la presencia de un virus de ARN. Las muestras se han recolectado y compartido libremente dentro de la comunidad de investigación mundial durante 13 años e incluyen todo, desde muestras de núcleos de hielo hasta estiércol animal.
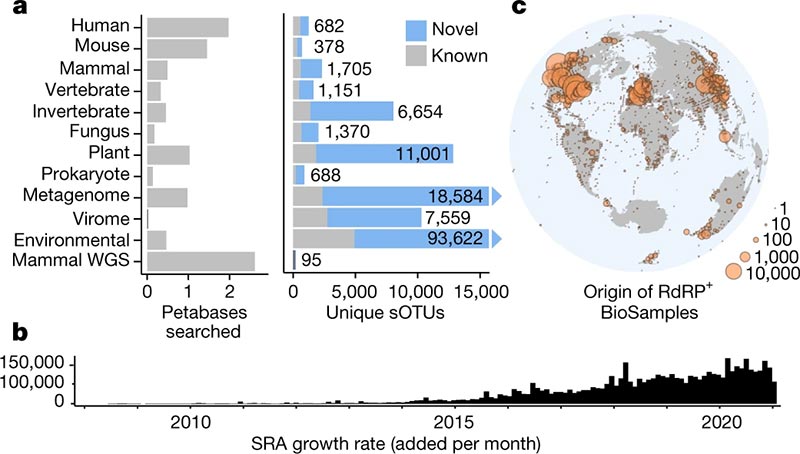
Tras este estudio ya se conocen 132.000 de estos virus
Los investigadores del Proyecto Serratus encontraron 132.000 virus de ARN (donde antes sólo se conocían 15.000) y nueve nuevas especies de coronavirus. Babaian estima que sin el CIC y la nube de AWS, un superordenador tradicional tardaría más de un año y cientos de miles de dólares en realizar los 2.000 años de tiempo de CPU necesarios para este análisis. Serratus lo logró en 11 días por 24.000 dólares.
«Estamos entrando en una nueva era de comprensión de la diversidad genética y espacial de los virus en la naturaleza, y cómo una amplia variedad de animales interactúa con estos virus. La esperanza es que no nos tome por sorpresa si algo como el SARS-CoV-2 —el nuevo coronavirus que causa COVID-19— emerge de nuevo. Estos virus se pueden reconocer más fácilmente y sus reservorios naturales se pueden encontrar más rápido. El verdadero objetivo es que estas infecciones se reconozcan tan temprano que nunca se conviertan en pandemias«, comenta Babaian, quien tiene un doctorado en genética médica de la UBC y ahora es miembro de Banting en la Universidad de Cambridge.
«Si un paciente presenta fiebre de origen desconocido, una vez que se secuencia la sangre, ahora puede conectar ese virus desconocido en el ser humano a una base de datos mucho más grande de virus existentes. Si un paciente, por ejemplo, presenta una infección viral de origen desconocido en St. Louis, ahora puede buscar a través de la base de datos en aproximadamente dos minutos y conectar ese virus a, digamos, un camello en el África subsahariana muestreado en 2012«, agrega Babaian.
El objetivo es que otra pandemia no nos coja por sorpresa
Babaian, de 32 años, estaba realizando una investigación genética sobre el cáncer con BC Cancer cuando llegó la pandemia de COVID-19 y cambió de objetivo. El trabajo, que el discreto Babaian asegura que comenzó como un «divertido proyecto paralelo«, comenzó el 3 de marzo de 2020, cuando él y su amigo y compañero de escalada, el estudiante de ingeniería de la UBC Jeff Taylor, esbozaron la idea «en el reverso de una servilleta«, según Babaian. «Debería haberme quedado con esa servilleta«, bromea.
Babaian se acercó al Centro de Innovación en la Nube de la UBC en busca de ayuda poco después. Así nació Serratus, que lleva el nombre de la montaña Serratus en la Cordillera de Tantalus en la Columbia Británica, que él y Taylor visitaron durante una escalada en 2020.
Babaian recordó que estaba sentado en la silla de enfermería de su esposa cuando los primeros resultados comenzaron a aparecer en su ordenador portátil, lo que indica que Serratus no sólo estaba calculando, sino que producía datos a una velocidad casi incomprensible.
Un momento emocionante
«Probablemente fue el período científico más emocionante de mi vida«, afirma. «Hay dos tipos de diversión. El primer tipo es de carácter sonriente y divertido. El segundo tipo lo experimentas es cuando te sientes miserable mientras lo haces, pero el recuerdo brilla, como escalar rocas. En muchos sentidos, Serratus es divertido en el segundo sentido. Simplemente tienes que creer que va a funcionar«.
Babaian afirma que no habría podido hacer este trabajo sin el apoyo del Centro de Innovación en la Nube de la UBC. «El Centro de innovación en la nube realmente estuvo allí para abrirnos las puertas«, señala. «Tuvimos una idea y trajeron a expertos de sus redes para hacerla realidad. Ahora la comunidad global puede beneficiarse de toda esta investigación que no se había explorado antes«.
«Artem se acercó a nosotros con una visión innovadora. El poder del Centro de innovación en la nube es que combinamos nuestros equipos internos de innovación y tecnología de UBC con los de Amazon Web Services«, indica Marianne Schroeder, directora del Centro de innovación en la nube de UBC. «Fue un gran privilegio apoyar la realización de esta visión; lo que hacemos es ayudar a encontrar una solución tecnológica para problemas complejos«.
Proyectos de código abierto y disponibles
El Centro, que se lanzó justo antes de la pandemia en enero de 2020, apoya desafíos que se enfocan en la salud y el bienestar de la comunidad. Hasta la fecha, el equipo ha publicado más de 20 proyectos que incluyen arquitectura de referencia y guías de implementación, todos de código abierto y disponibles.
«Si bien la nube pública tal como la conocemos ha existido durante 15 años, los últimos años de innovación en Amazon Web Services realmente han hecho posible la investigación genómica de una nueva manera”, comenta Coral Kennett, quien dirige el Centro de Amazon Web Services. “Pudimos darle a Artem acceso al poder de computación a centavos por consulta. Alentamos encarecidamente a la comunidad de investigación a enviar sus proyectos e ideas al Centro de innovación en la nube para que salgan a la luz más innovaciones que beneficien a la comunidad«.
Fuente: Nature.